参考基因组介绍
参考基因组(reference genome)
人类基因组计划绘制了人类的基因图谱,得到了30亿个碱基对的序列,投入30亿美元,使用第一代测序技术,将测序得到的序列组装(拼接/装配/Assembly)。这个序列也成为最初的参考基因组。
理解测序原理是一个难点,它也很重要,不然后面会有好多不明白的地方。这里推荐一篇很好的博文,三代基因组测序技术原理简介,感谢原作者。
推荐两个YouTube上的视频,可以直观的感受一下测序技术。
谷歌,百度,知网,PubMed等可以用来获取更多信息,搜索关键字可为:DNA测序,测序技术等。
前两天总结了 一些提高效率的系统调整,现在总结一些常用的提高效率的工具。
通常,新机装好,我会装上一个小软件,那就是Notepad++,包括现在我在Windows下面写笔记也是用的这个,对于任何文本文件,它都可以方便的右键打开。如果是开大文件,EmEditor是个不错的选择。当然如果系统中存在一个bash终端或者有Git GUI类似可以使用Linux强大命令工具,vim/more/less可以随便用了。 
1 | $ cat goagent-gtk.desktop |

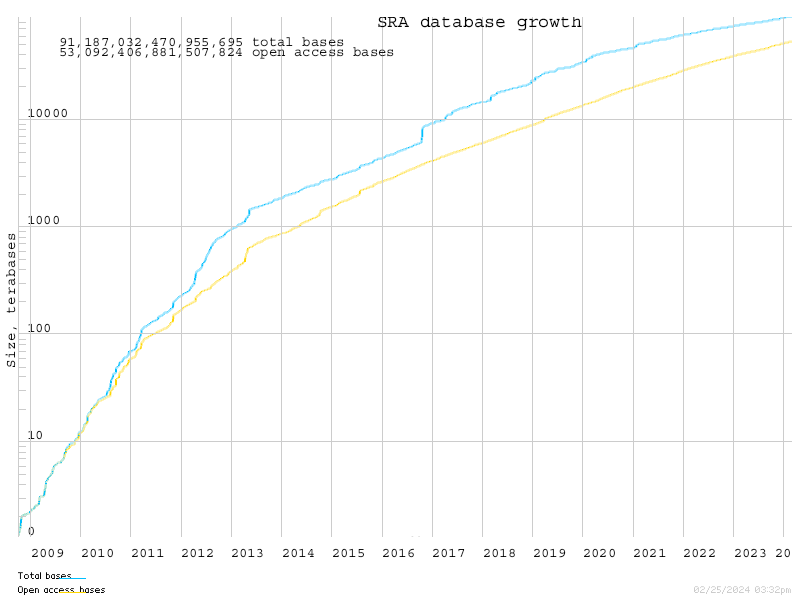
这里主要介绍NCBI的SRA和EBI的ENA,两者同步,数据基本相符,有的study在ENA中有BAM文件,但SRA中没有。网址分别是
ENA: http://www.ebi.ac.uk/ena
SRA: http://www.ncbi.nlm.nih.gov/sra